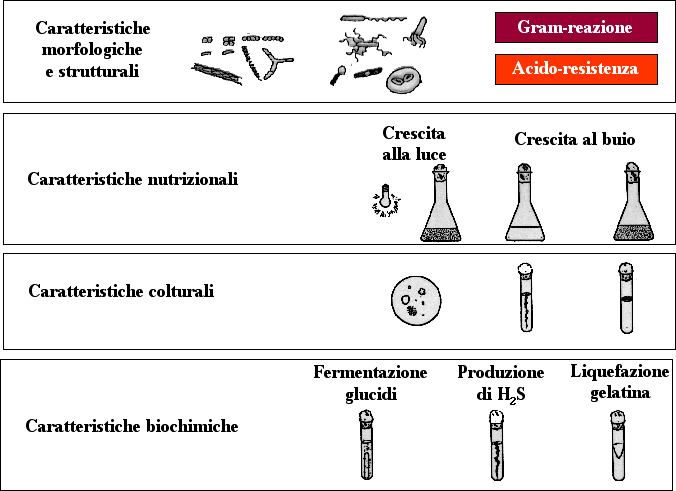
8. Identificazione dei microrganismi
Da un punto di vista applicativo, l'identificazione dei microrganismi è importante perché consente di accoppiare un nome ad un insieme di proprietà possedute da un'entità microbica rinvenibile in un determinato habitat.
Da un punto di vista scientifico, l'identificazione è importante per lo scambio, il confronto, la catalogazione, l'archiviazione e l'unificazione delle conoscenze.
I microrganismi possono essere identificati, come specie già note o come nuove entità tassonomiche, sulla base di caratteristiche fenotipiche e/o genotipiche.
Di seguito sono ricordate le caratteristiche fenotipiche di rilevanza tassonomica.
Va ricordato che esse, quale risultato dell'espressione genica, devono essere determinate in condizioni ben definite.

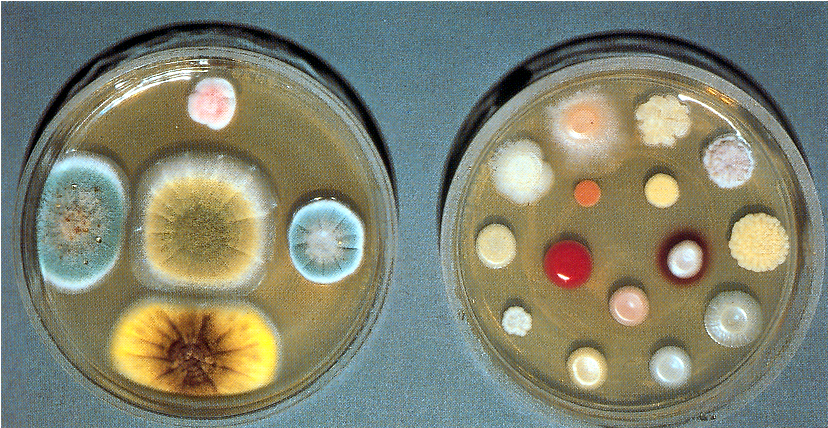
Già l'aspetto della colonia che si sviluppa sulla piastra di isolamento può essere di rilevanza diagnostica. Questo carattere può infatti essere definito per ogni specie microbica in determinate condizioni colturali.
Nella figura che segue sono riportate colonie microbiche ben diverse fra loro.
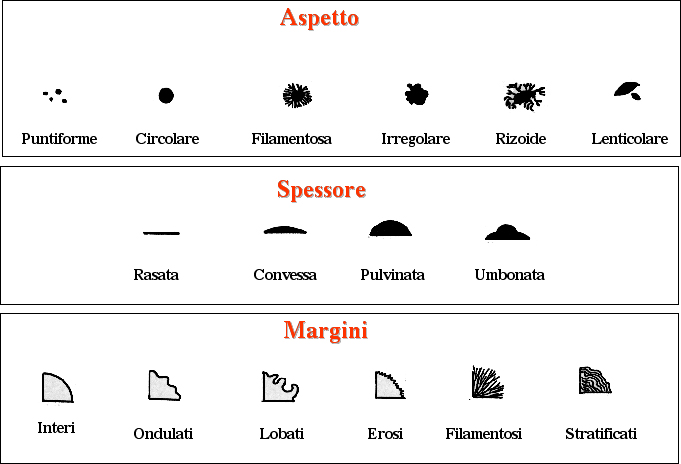
L'aspetto, lo spessore ed i margini delle colonie sono descritti ricorrendo agli attributi riferiti nella figura che segue.
Analogamente, sono rilevanti le
modalità di crescita su agar "a becco di clarino", in brodo ed
in tubi solidificati per intero con gelatina. In quest'ultimo caso si
può avere o non fluidificazione del mezzo per attività
proteolitica; e, quando ricorre, è caratteristica anche la
modalità di detta fluidificazione.
I vari casi che si possono riscontrare sono schematizzati,
nell'ordine di citazione, nelle tre figure che seguono.



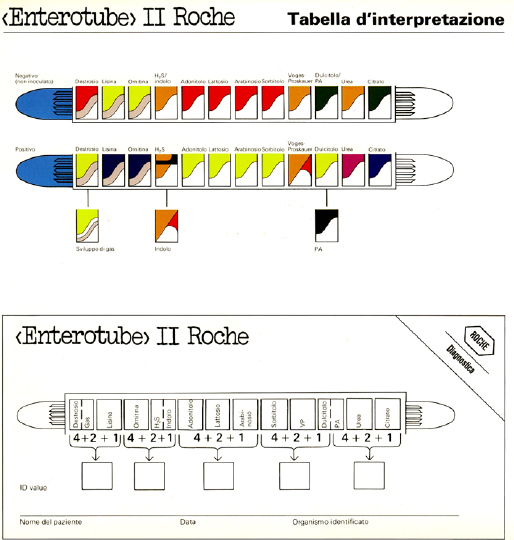
La determinazione delle
caratteristiche biochimiche di un microrganismo costituisce un lavoro
che può risultare dispendioso e laborioso, la cui
utilità dipende anche dal numero delle caratteristiche che
vengono considerate. Pertanto, sono stati messi a punto sistemi
miniaturizzati ed informatizzati di elevata praticità ed in
grado di agevolare molto l'identificazione dei microrganismi quando
le colture in studio possono già essere riferite a specifici
raggruppamenti sulla base di determinazioni preliminari di altro
tipo.
Le quattro figure che seguono ricordano alcuni dei suddetti
sistemi.
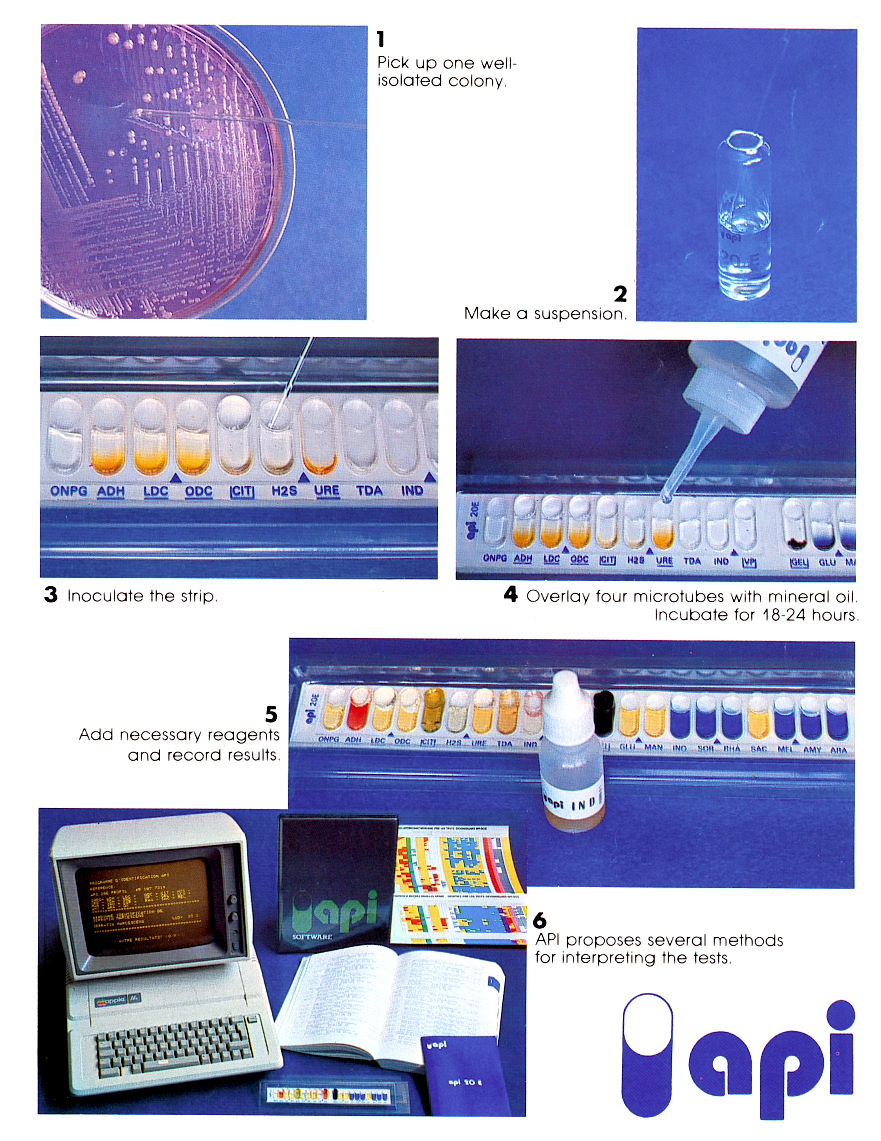


Altre caratteristiche di rilevanza diagnostica sono quelle sierologiche e quelle di patogenicità, ricordate nella figura che segue.

Ma informazioni molto precise sulla natura dei microrganismi, come di tutti i viventi, possono oggi essere ricavate dall'analisi del loro genoma.
La suggestiva figura che segue ne ricorda le potenzialità.

La biotipizzazione degli isolati microbici attraverso le caratteristiche genotipiche può essere applicata al conseguimento di diverse finalità, come schematizzato nella figura che segue.

Fra i caratteri genotipici, il primo, storicamente, è quello relativo alla composizione in basi del DNA: % G+C.
Si tratta di un dato piuttosto grossolano, ma sicuramente significativo per ogni gruppo tassonomico di microrganismi.
Di seguito è ricordata una delle tecniche che consentono la determinazione della percentuale di G+C di un DNA attraverso lo studio della denaturazione termica.

Con esperimenti di questo tipo, il G+C% viene ricavato dalla formula:
Tm = 69,3 + 0,41 (% G+C)
Può essere utile ricordare le relazioni riportate nel riquadro che segue.

Un'altra tecnica fondamentale nell'accertamento della identità di una coltura microbica è l'ibridazione DNA-DNA.
La figura che segue ricorda che il DNA a doppia elica si separa in due filamenti a temperature superiori alla Tm, per tornare alla condizione nativa con il successivo raffreddamento.

Se si denatura termicamente una
miscela di DNA estratti da due colture della stessa specie, marcati
però con due radioisotopi diversi (per esempio
14C
e
3H),
con la rinaturazione si ottengono doppie eliche "ibride" con doppia
radioattività.
Ciò si verifica perché in questo caso esisterà
sicuramente 100% di omologia fra i due DNA.
Ma in alternativa al DNA totale,
esistono regioni genomiche particolarmente significative,
relativamente alla individualità di un organismo vivente. Sono
gli "operoni" che contengono i geni che codificano per gli RNA
ribosomiali (nei procarioti: 16 S, 23 S e 5 S).
Si tratta di regioni "ben conservate", in quanto recanti informazioni
che riguardano elementi essenziali della cellula,quali i ribosomi:
elementi evoluzionisticamente ottimizzati.
La figura che segue schematizza la struttura dell'rDNA, mostrando le parti altamente "conservate" e quelle più o meno variabili, fiancheggianti o interposte, cioè spaziatrici.

L'rRNA 16 S riveste una
grandissima importanza nella diagnostica speciografica dei
procarioti.
Si tratta di un filamento singolo di RNA composto da 1.500
ribonucleotidi, con porzioni appaiate, in sequenza, come già
detto, "altamente conservata", ma con variazioni specifiche per
ciascun
taxon.
Nella figura che segue è riportata la struttura dell'rRNA 16 S di Escherichia coli.

In queste molecole sono state individuate nove regioni variabili, di qualche centinaia di basi, indicate da V 1 a V 9, rilevabili nello schema che segue.

Le regioni V possono essere
amplificate mediante PCR.
Ogni prodotto della PCR può essere sequenziato e la sequenza
determinata può essere comparata con quelle (ormai numerose)
presenti in banche-dati genetiche. L'esito del confronto, di solito
corredato di percentuale di omologia, può essere utile per
identificare il microrganismo.
Ampliconi di identica dimensione ma con sequenza diversa possono essere separati per elettroforesi in gradiente denaturante di temperatura (TGGE) o di forza ionica (DGGE).
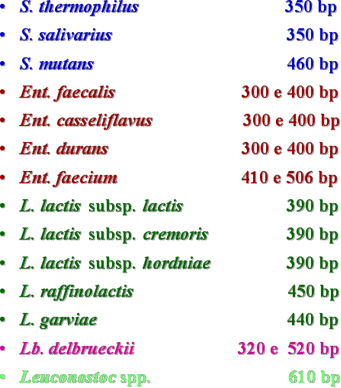
Nelle figure che seguono sono riportati i comportamenti delle regioni V 3 del 16 S rDNA di batteri lattici e di micro-stafilococchi.

a = Pediococcus acidilactici DSM 20333
b = Pediococcus pentosaceus DSM 20336
c = Enterococcus casseliflavus ATCC 25788
d = Enterococcus durans ATCC 11576
e = Enterococcus faecalis ATCC 19433
f = Enterococcus faecium ATCC 19434
g = Lactobacillus sakei DSM 20017
h = Lactobacillus curvatus LTH 1432
i = Lactobacillus casei ATCC 334
j = Lactobacillus delbrueckii subsp. lactis DSM 20072
k = Lactobacillus delbrueckii subsp. bulgaricus DSM 20081
l = Lactobacillus helveticus ATCC 15009
m = Lactobacillus plantarum NCDO 1193
n = Leuconostoc mesenteroides subsp. mesenteroides DSM 20343
o = Lactococcus lactis subsp. lactis DSM 20481
p = Lactococcus lactis subsp. cremoris DSM 4645
q = Lactococcus lactis subsp. hordniae DSM 20450
r = Lactococcus garvieae DSM 20684
s = Leuconostoc lactis DSM 20202
t = Weissella paramesenteroides DSM 20288
u = Streptococcus thermophilus CNRZ 302

a = Staphylococcus xylosus DSM 20266, S. warneri DSM 20316
b = S. capitis subsp. capitis DSM 20326, S. hominis subsp. hominis DSM 20328
c = S. lentus DSM 20352, S. carnosus subsp. carnosus DSM 20501
d = S. schleiferi subsp schleiferi DSM 4807, S. epidermidis DSM 1798
e = S. intermedius DSM 20373, S. kloosii DSM 20676
f = S. chromogenes DSM 20454, S. caprae DSM 20608
g = S. auricularis DSM 20609, Micrococcus flavus DSM 1790
h = S. aureus subsp. aureus DSM 20231, S. haemolyticus DSM 20263
i = S. epidermidis DSM 20044, S. saprophyticus subsp saprophyticus DSM 20229
j = S. gallinarum DSM 20610, S. condimenti DSM 11674
k = S. sciuri subsp. sciuri DSM 20345, S. lugdunensis DSM 4804
l = S. cohnii subsp. cohnii DSM 20260, S. equorum DSM 20674
m = S. simulans DSM 20322
M = Marker 1Kb DNA ladder plus (Gibco-BRL).
Se si estrae il DNA totale da una qualsiasi matrice, si amplifica una delle regioni V impiegando primer ricavati dall'rDNA di E. coli e si separano gli ampliconi per DGGE o TGGE, si ottengono pattern mostranti una banda per pressoché ogni specie eubatterica presente, molto adatti a fornire un'idea della biodiversità batterica che caratterizza l'habitat analizzato.
Nella figura che segue sono riportati i pattern forniti dall'analisi PCR-DGGE relativa alla regione V 3 dell'rDNA 16 S di diversi formaggi.

Informazioni di grande utilità diagnostica e tassonomica possono essere ricavate anche dalla spacer analysis.
Dall'amplificazione della regione
spaziatrice 16 S-23 S si evidenzia che specie o generi diversi
possiedono spacer di dimensioni diverse. Alcuni mostrano più
classi di spacer, in quanto dotati di più operoni rDNA.
Di seguito sono riportati gli spacer di alcuni microrganismi di
interesse alimentare e di alcuni formaggi.

Dimensioni degli
"spacer" di Batteri lattici
Profili "Spacer" di
alcuni prodotti commerciali

Porzioni dell'rDNA, come porzioni
più o meno grandi di geni specie-specifici, ma anche altre
sequenze individuabili con vari metodi, sono usate per la messa a
punto di "sonde" oligonucleotidiche atte alla individuazione di
determinati taxa.
Le stesse sequenze consentono l'identificazione attraverso PCR.
Nella figura che segue appare quanto risultato dall'uso di una sequenza ricavata dai profili RAPD (di cui si dirà fra breve), di ceppi di Leuconostoc mesenteroides subsp. mesenteroides.

Di seguito si possono rilevare le sequenze utilizzate per sonde atte ad individuare batteri lattici.

Per discriminare ceppi o biotipi
nell'ambito di isolati riferibili alla stessa specie si possono
utilizzare diversi approcci analitici.
Particolarmente valido è il "DNA-fingerprinting".
Esso viene ottenuto digerendo il DNA con una endonucleasi di
restrizione e separando i frammenti per elettroforesi.
I frammenti di DNA sono evidenziati alla luce UV, rendendoli
fluorescenti con bromuro di etidio, una sostanza che si va a
intercalare fra le due doppie eliche del DNA, come mostrato nella
figura che segue.

Se si utilizza una endonucleasi di restrizione che "taglia" molto, producendo molti piccoli frammenti, questi possono essere separati per elettroforesi convenzionale.
La figura che segue mostra il fingerprinting ottenuto per analisi di restrizione (REA) mediante Hind III del DNA di ceppi di Leuconostoc mesenteroides.
Se si adopera un'endonucleasi che taglia più raramente, producendo pochi grossi frammenti, questi vanno separati mediante elettroforesi in campo pulsato.
Di seguito sono riportati DNA-fingerprinting ottenuti mediante PFGE (Pulsed Field Gel Electrophoresis).



Ma esistono diversi altri metodi
di biotipizzazione che permettono di distinguere autentici "ceppi" o
"biotipi" fra le diverse colture isolate.
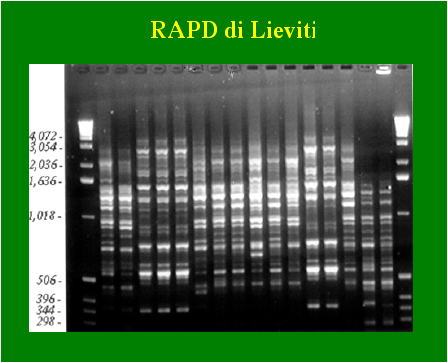
Molto usata è una tecnica che ricorre alla PCR: la "Random
Amplified Polymorphic DNA Analysis", nota come RAPD.
Di seguito sono riportate RAPD di Batteri lattici e di Lieviti.

Ma un fingerprinting può essere ottenuto anche dall'elettroforesi delle proteine totali cellulari.
La figura che segue ne mostra un esempio.

Un'altra maniera per differenziare vari ceppi microbici è quella che si serve della determinazione della loro resistenza a diversi antibiotici. In questo caso si parla di antibiotyping.
Nella tabella che segue, riportante dati relativi a ceppi di Staphylococcus xylosus, si può rilevare che spesso si riscontra corrispondenza fra questo criterio di raggruppamento dei ceppi ed altri mostrati in precedenza.

Con l'avvento dei computer, i dati relativi alla caratterizzazione degli isolati microbici, quanto più numerosi, e sia fenotipici che genotipici, possono utilmente essere impiegati per un approccio numerico alla tassonomia. In questo caso si parla di TASSONOMIA NUMERICA.
Le figure successive ricordano le procedure della tassonomia numerica e le modalità di rappresentazione ed utilizzazione dei risultati.




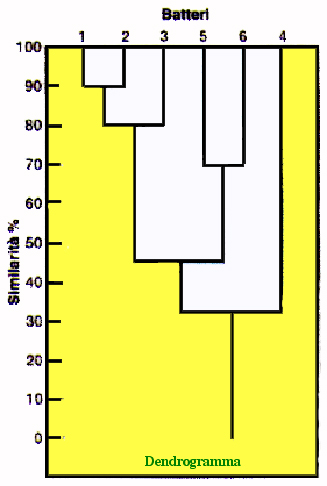
L'applicazione della cluster analysis porta all'individuazione di gruppi (clusters) di similarità e può risultare molto utile, anche nelle biotecnologie microbiche alimentari.
Nella figura che segue è riportato il risultato della cluster analysis applicata ai profili RAPD di un gran numero di ceppi di streptococchi lattici isolati da vari habitat.

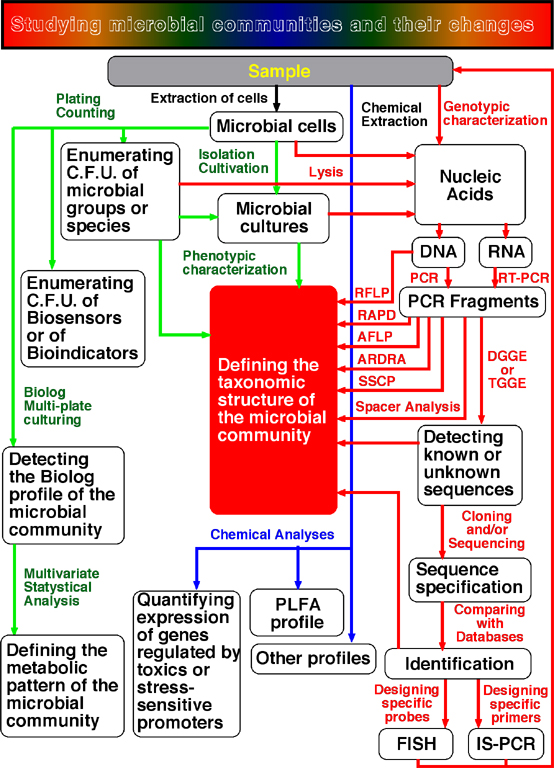
Di tutte le tecniche di caratterizzazione dei microrganismi che sono state ricordate, diverse possono essere applicate, da sole od insieme ad altre, mediante un approccio cosiddetto "polifasico", per rilevare o monitorare, nella loro struttura di specie e di biotipi, le comunità microbiche ricorrenti sia negli ecosistemi naturali sia in quelli tecnologici.
La figura che segue rappresenta un tentativo di ricapitolazione generale delle principali procedure che possono essere seguite nello studio della struttura delle popolazioni microbiche.
Torna all'indice
della
GuidaVai alla parte
nona